| 所属 |
① 横浜市立大学 大学院生命医科学研究科 ② 横浜市立大学 大学院生命医科学研究科 |
|
|---|---|---|
| 氏名 |
① 西村 善文 ② 栗田 順一 |
|
| AMED 事業 |
課題名 | 生命科学と創薬研究に向けた相関構造解析プラットフォームによる支援と高度化 |
| 代表機関 | 理化学研究所 | |
| 代表者 | 山本 雅貴 | |
NMRスクリーニング、19F-NMR、リアルタイムモニタリング、天然変性タンパク質、LC-NMR
当機関では800MHzという高磁場においてNMRスクリーニングを可能とする多検体自動測定装置を導入して1H,13C,15Nの核種について測定可能です。また、700MHzでは19Fも測定可能な他検体自動測定装置も導入しています。
低分子化合物による化学シフトの変化の観測は測定法の選択や試料の調製などにノウハウを要します。当機関では、試料の調製やその観測法の選択などの支援を行いますので円滑に結果を得られます。
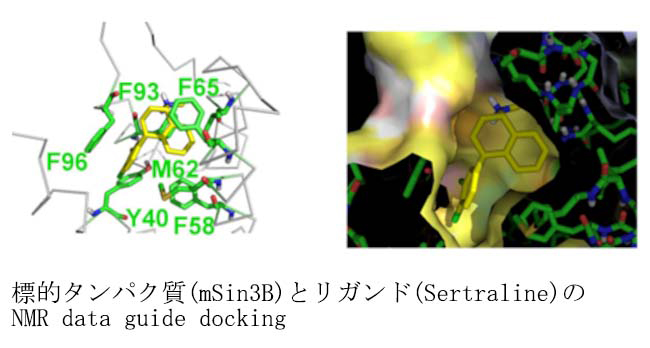
スクリーニングの初期において、リガンドと基質の結合も弱い段階では、直接的な結合情報を得ることが難しいことがあります。当機関では、化学シフト変化や水素交換速度の変化などの結合が弱くとも得られる相互作用情報から、ドッキングシミュレーションにて複合体構造を得る構造解析支援を行っております。また、ドッキングシミュレーションで得られた複合体の構造の評価をNMRのスピン拡散を使いその構造のもっともらしさを評価することも支援できます。
薬物動態にはLC-MSなどが非常に強力なツールとして使われておりますが、MSだけの情報で化合物を同定することが困難であることも多く、当機関では950MHzという超高磁場に世界で初めてLCシステムを連動させ微量化合物の同定を可能にしました。また700MHzのLC-NMRも使用可能です。
天然変性領域の動的な相互作用変化をNMRでは検出することが可能です。
DNAおよびRNAの結合によるタンパク質の化学シフト変化の観測は測定法の選択や特にその試料の調製などにノウハウが必要です。当機関では、試料の調製やその観測法の選択や結合部位の同定の解析支援を行います。
RNAやDNAの構造解析は、タンパク質と比べ安定同位体標識が困難である上にシグナルの重なりが多いため特に経験が必要となりますが、今までのBINDSの経験等からの構造解析を通してメチル化などの化学修飾を用いた構造解析のノウハウが蓄積されております。またNMR用の核酸特異的な安定同位体ラベルの経験もあります。
NMRでは残基ごとに信号が得られるため、NMR試料管内で酵素反応を行うことにより、残基のシグナル変化を経時的に追うことができ反応速度と反応部位の同定が可能です。当機関では特にリン酸化のリアルタイムモニタリングや反応部位の同定に豊富な経験があります。
図に示すように当機関では光ファイバーによりNMRサンプル内に光を引き込んだ測定などの装置構築、測定の豊富な経験がありますので光NMRによる構造解析支援が可能です。

ヒトは約300種類の細胞からなるが、各細胞は基本的には同じDNAを持っている。同じDNAをもっていても細胞機能が異なるのはDNAの高次構造であるクロマチン構造が異なるためである。この現象をエピゲノムと呼ぶ。クロマチンの構造単位は4種類のヒストン(H2A,H2B,H3,H4)各2個で形成されるヒストン8量体に約146塩基対のDNAが巻き付いたヌクレオソームである。ヌクレオソームのコア構造から突き出した天然変性状態にある各ヒストンの末端テール領域のアセチル化、メチル化、リン酸化、ユビキチン化等でクロマチン構造は変化し、遺伝子発現等が制御される。クロマチン構造に異常が生じると、癌や神経疾患をはじめ様々な疾病が発症する。これらヒストンテールは天然変性領域で結晶構造解析やクライオ電顕構造では同定できない。NMRではこれらテールのアミノ酸の動的構造を解析でき、テール領域のアセチル化、メチル化、リン酸化、ユビキチン化による動的な相互作用変化を同定する事が可能である。我々はNMRを用いてヌクレオソーム上でのヒストンテールのネットワークを初めて解析し報告した。
クロマチン構造には遺伝子の発現が促進されているユークロマチンと抑制されているヘテロクロマチンがある。ユークロマチンでは転写開始部位のヌクレオソームはクロマチンリモデリング因子等によりはがされ、むき出しになったDNAに基本転写因子とRNAポリメラーゼが結合しRNAを合成していく。RNA合成時にはヒストンシャペロンやクロマチンリモデリング因子によりヌクレオソームの構造が連続的に変化していく。
セントロメアやテロメアに代表されるヘテロクロマチンではヒストンH3の9番目のリシンがメチル化されヘテロクロマチンタンパク質のHP1が結合し、凝集したクロマチンを形成する。またヌクレオソームを結合しているリンカーDNAにはヒストンH1が結合する。我々は、ヒストンH1が結合したクロマトソームのNMR構造も解析し、ヌクレオソーム中のヒストンテールとの動的構造との違いも報告した。またヘテロクロマチンタンパク質HP1とメチル化ヒストンH3の相互作用や細胞周期的なパッセンジャー複合体との相互作用、ヒストンシャペロンNAP1やFACTとヒストンH2AH2BやH3H4との相互作用、テロメア形成タンパク質TRF2と4重鎖DNAとの相互作用等をNMRにより解析している。
特に、NMRによりヌクレオソーム中でヒストンH4のテールのアセチル化により、ヒストンH3のテールの動的構造が変化し、更にアセチル化されやすくなることを始めて見出し、ヌクレオソーム上でのヒストンテールのネットワーク機構を解明し報告した。またNMRを用いて基本転写/修復因子TFIIHによる基本転写因子TFIIE、がん抑制因子p53、DNA修復因子XPCやUVSSA、細胞周期特異的転写因子DP1、3種類のRNAポリメラーゼ等のリクルート機構を解明してきた。
これら核内タンパク質のテールやヒンジやリンカーの天然変性領域が、伸びた紐状構造で標的タンパク質と結合したり、あるいは紐状構造同士で動的に絡み合ってタンパク質間相互作用に寄与しているが、伸びた紐状構造はX線結晶構造解析やクライオ電顕構造解析では同定出来ない。紐状構造の動的な構造解析としてNMRが必須である。巨大タンパク質のTFIIHのクライオ電顕構造中で揺らいで見えていないp62サブユニットのPHドメインの揺らぎ構造をNMRで解析し、NMR構造をクライオ電顕構造にドッキングする事で、TFIIHがTFIIEやp53やXPCやUVSSAをリクルートする機構をTFIIHの全体構造中で解明できた。また3種類のRNAポリメラーゼのPolI、PolII、PolIIの共通サブユニットRpb6のN末テールは天然変性領域でクライオ電顕構造中では同定されていなかったが、TFIIHのリクルートに関与する事を見出した。Rpb6のN末テールとTFIIHのリクルート構造のNMR構造に基づいて、PolIIのクライオ電顕構造とTFIIHのクライオ電顕構造をドッキングする事に成功し、複合体全体構造の描像を得ることが可能となった。
このような天然変性領域と標的タンパク質間相互作用解析技術の創薬への応用として膠芽腫や神経疼痛に関連する神経特異的転写因子NRSF/RESTの機能を阻害する化合物を同定し、神経疾患治療薬物の開発も目指している。実際に線維筋痛症のモデルマスを用いた実験で効果が示されている。
さらに光応答タンパク質の暗状態と光照射状態の構造変化を解析するために、光を照射しながらNMRを測定できる光NMRをセットし、解析を進めている。