| 所属 |
① 東北大学 大学院情報科学研究科・東北メディカル・メガバンク機構 ② 東北大学 未来型医療創成センター・東北メディカル・メガバンク機構 ③ 東北大学 大学院情報科学研究科・東北メディカル・メガバンク機構 |
|
|---|---|---|
| 氏名 |
① 木下 賢吾 ② 勝岡 史城 ③ 西 羽美 |
|
| AMED 事業 |
課題名 | マルチオミックス・ヒューマンバイオロジー解析基盤の高度化と支援 |
| 代表機関 | 東北大学 | |
| 代表者 | 木下 賢吾 | |
ヒューマンバイオロジー、一般住民ゲノムコホート、全ゲノム解析、scRNA-seq, メタボローム
本事業では、ヒト、特に日本人集団に特化した(a)ゲノム解析、(b)メタボローム解析、(c)in-vitro層別化代謝評価、及び(d)ヒト細胞ライブラリ提供を中心に、その他(e)一細胞トランスクリプトーム、一細胞ゲノム解析、メチローム、ChIP-seqも組み合わせた最先端解析支援を行う。ヒト対象研究では倫理面、情報セキュリティ対策等での特別な配慮が必要だと言う理由だけでなく、対照群データの取得が難しいという課題がある。これに対して本事業では、東北メディカル・メガバンク機構(ToMMo)がこれまで整備してきた一般住民の解析データと解析基盤及び、現BINDSで開発を行ってきた手法やデータベースを活用し、独自性の高い支援環境を提供する。
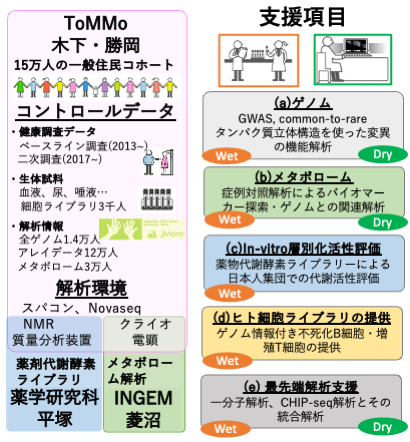
各項目の詳細は以下の通りである。なお、(b)と(c)に関しては、支援メニューの別項目としても挙げている。
(a) 全ゲノム解析に関しては情報解析に特化した支援を行う。個別支援で解析する全ゲノム情報に対しては、ToMMoで解析を実施した一般住民の全ゲノム情報(現時点で1.4万人、最終的には10万人規模)を対照群とする統合解析を支援する。アレイデータに対する遺伝子型インピュテーションやToMMoの詳細な表現型を用いたGWAS解析も支援する。なお、後述する高度化対象のヒトゲノム難読領域(HLA及びCYPなど)に関するターゲットシークエンスに関しては、ウェット解析支援も行う。(b) メタボローム解析に関しては、ヒト由来の血漿、尿、細胞試料に対するNMR装置または質量分析装置での解析を支援する。特に、ヒト血漿サンプルに対する解析では、既に4万検体を対照群として、ToMMoの解析手法と同じ解析プロトコルを用いることで、詳細な表現型とゲノム情報の比較も含めた高度な統合解析を提供する。ヒトとの比較が可能な研究計画の場合には、一部マウス由来試料の解析も支援する。(c) In-vitro層別化代謝評価(薬物代謝酵素ライブラリによる日本人集団での薬物代謝多様性測定)に関しては、分担研究者の平塚らが、AMEDゲノム創薬基盤推進研究事業の支援を受けて独自に構築を行った「代表的な薬物代謝酵素16種に対して、ToMMo8.3KJPNで同定された705変異を導入したタンパク質ライブラリ」を活用し、支援希望者が提供する低分子の日本人集団における代謝変化多様性を定量化し、創薬展開の前段階としてのin-vitro代謝評価を支援する。(d) ヒューマンバイオロージーの課題の一つとして、薬剤等への外的な刺激への応答の解析を生体で行うことが難しい点がある。そこで本事業では、ToMMoでこれまで樹立した全ゲノム情報付きの3000人分を超えるヒト細胞ライブラリ(不死化B細胞と増殖T細胞)を機能解析に提供し、培養細胞によるコホート(Cohort in a dish)の実施を支援する。これら細胞は全てToMMoのコホート参加者由来であり、全ゲノム解析が終了しているだけでなく、詳細な健康調査情報を経時的に取得しており、特定の変異に着目した機能解析が可能な点が世界に類を見ないライブラリである。(e)以上4つを主な支援とするが、一細胞ゲノム解析によるCNVの同定、一細胞トランスクリプトームによる細胞分化の解析や、細胞周期を同調させたCUT&Tag解析など最先端のゲノム・オミックス解析を多数した実績をベースに、厳選した課題に対して高度な情報解析とセットでの支援を実施する。
・ポストGWAS解析をタンパク質の立体構造情報などを活用し実施した例
Yamada M, Motoike IN, Kojima K, Fuse N, Hozawa A, Kuriyama S, Katsuoka F, Tadaka S, Shirota M, Sakurai M, Nakamura T, Hamanaka Y, Suzuki K, Sugawara J, Ogishima S, Uruno A, Kodama EN, Fujino N, Numakura T, Ichikawa T, Mitsune A, Ohe T, Kinoshita K, Ichinose M, Sugiura H, Yamamoto M. Genetic loci for lung function in Japanese adults with adjustment for exhaled nitric oxide levels as airway inflammation indicator. Commun Biol 4(1):1288. 2021 doi: 10.1038/s42003-021-02813-8.
Shido K, Kojima K , Shirota M, Yamasaki K, Motoike IN, Hozawa A, Ogishima S, Minegishi N, Tanno K , Katsuoka F, Tamiya G, Aiba S, Yamamoto M, Kinoshita K. GWAS Identified IL4R and the Major Histocompatibility Complex Region as the Associated Loci of Total Serum IgE Levels in 9,260 Japanese Individuals. J. Invest Derm. 141(11):2749-2752. 2021. doi: 10.1016/j.jid.2021.02.762
・メタボローム解析例
Saigusa D, Hishinuma E, Matsukawa N, Takahashi M, Inoue J, Tadaka S, Motoike I N, Hozawa A, Izumi Y, Bamba T, Kinoshita K, Ekroos K, Koshiba S, Yamamoto M. Comparison of kit-based metabolomics with other methodologies in a large-cohort, towards establishing reference values. Metabolites, 11: 652.2021. doi: 10.3390/metabo11100652.
・マルチオミックスでの統合解析例
Okazaki K, Anzawa H, Katsuoka F, Kinoshita K, Sekine H, Motohashi H. CEBPB is Required for NRF2-Mediated Drug Resistance in NRF2-Activated Non-Small Cell Lung Cancer Cells . J Biochem 171(5):567-578. 2022. doi: 10.1093/jb/mvac013.
・薬物代謝酵素に日本人集団で見られる低頻度変異の機能解析例
Kumondai M, Gutierrez Rico EM, Hishinuma E, Nakanishi Y, Yamazaki S, Ueda A, Saito S, Tadaka S, Kinoshita K, Saigusa D, Nakayoshi T, Oda A, Hirasawa N, Hiratsuka M. Functional Characterization of 21 Rare Allelic CYP1A2 Variants Identified in a Population of 4773 Japanese Individuals by Assessing Phenacetin O-Deethylation. J Pers Med 11(8):690. 2021. doi: 10.3390/jpm11080690.
代表者の研究概要について:http://www.sb.ecei.tohoku.ac.jp/
東北メディカル・メガバンク計画について:https://www.megabank.tohoku.ac.jp/
日本人マルチオミックス参照データベース:https://jmorp.megabank.tohoku.ac.jp/