| 所属 |
① 東北大学 大学院情報科学研究科 ② 東北大学 大学院医学系研究科 |
|
|---|---|---|
| 氏名 |
① 川端 猛 ② 城田 松之 |
|
| AMED 事業 |
課題名 | マルチオミックス・ヒューマンバイオロジー解析基盤の高度化と支援 |
| 代表機関 | 東北大学 | |
| 代表者 | 木下 賢吾 | |
分子モデリング、ホモロジー・モデリング、複合体モデリング、変異影響評価、AlphaFold2
本事業では、タンパク質立体構造へのゲノム変異マッピングの高度化を行っている。そのために、PDBの実験構造だけではなくAlphaFold2などの高精度予測構造も積極的に利用して、タンパク質単量体、タンパク質複合体、化合物-タンパク質複合体、核酸-タンパク質複合体など多様な立体構造モデルを作成するためのプログラム開発、WEBサーバ開発を行っている。これらの技術を活用することで、様々なタンパク質の立体構造モデルを作成し、変異部位のマッピング、変異の影響の評価、人工変異導入の設計などに供する研究支援を行う。
・タンパク質の立体構造の予測・モデリング
・タンパク質複合体、低分子化合物-タンパク質複合体の予測・モデリング
・変異部位の立体構造へのマッピング
・立体構造を用いたアミノ酸変異の影響評価
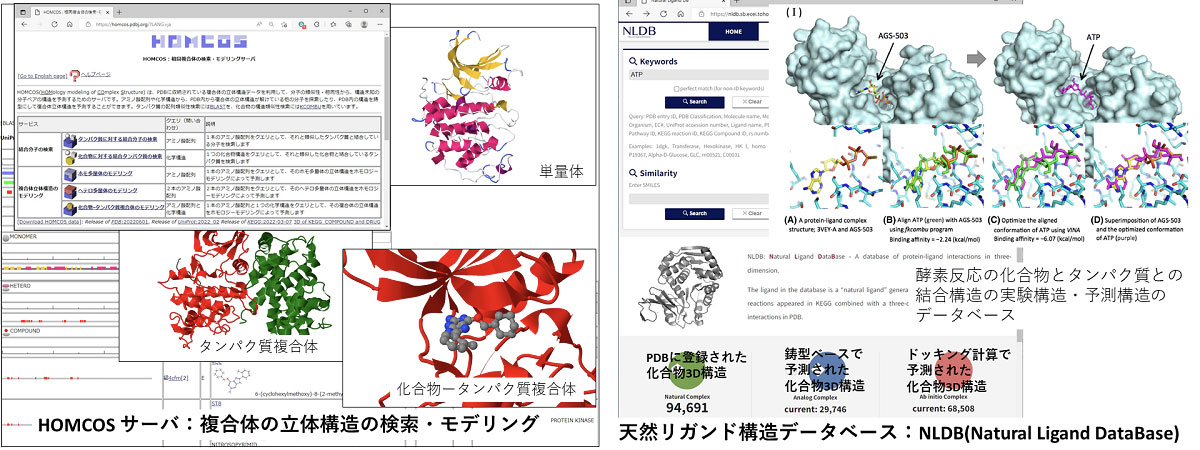
・複合体検索・モデリングサーバHOMCOSを利用した支援
https://homcos.pdbj.org
・天然リガンド立体構造データベースNatural Ligand Database NLDBを利用した支援
https://nldb.sb.ecei.tohoku.ac.jp/nldb/top
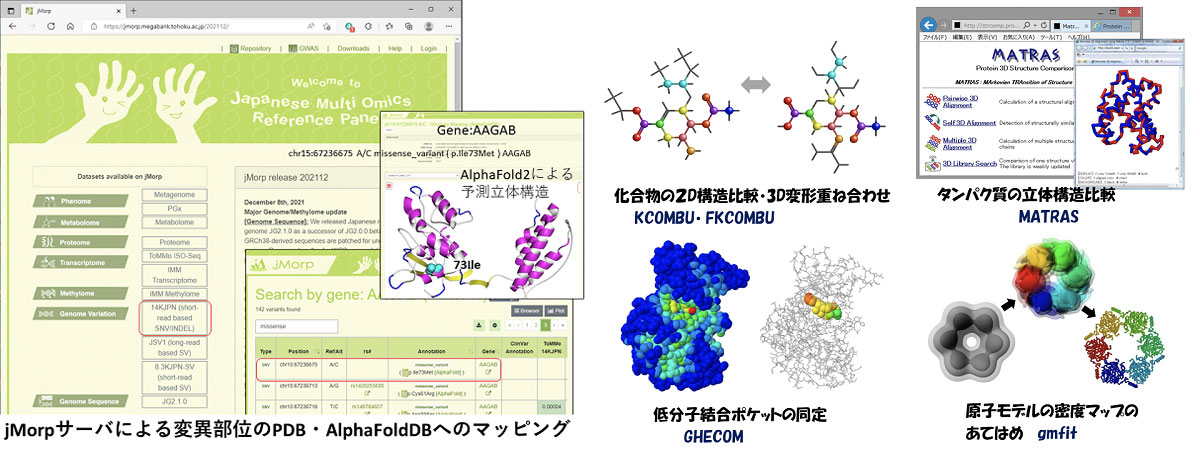
・jMorpサーバによる変異部位のPDB・AlphaFoldDBへのマッピング
https://jmorp.megabank.tohoku.ac.jp/202112/
・化学構造比較・重ね合わせプログラムKCOMBUを用いた支援
https://pdbj.org/kcombu/
・結合ポケット同定プログラムghecomを用いた支援
https://pdbj.org/ghecom/
・電顕マップ・原子モデル重ね合わせツールgmfitを利用した電顕密度マップからの原子モデリング支援
https://pdbj.org/gmfit/
・立体構造比較プログラムMATRASを用いた支援
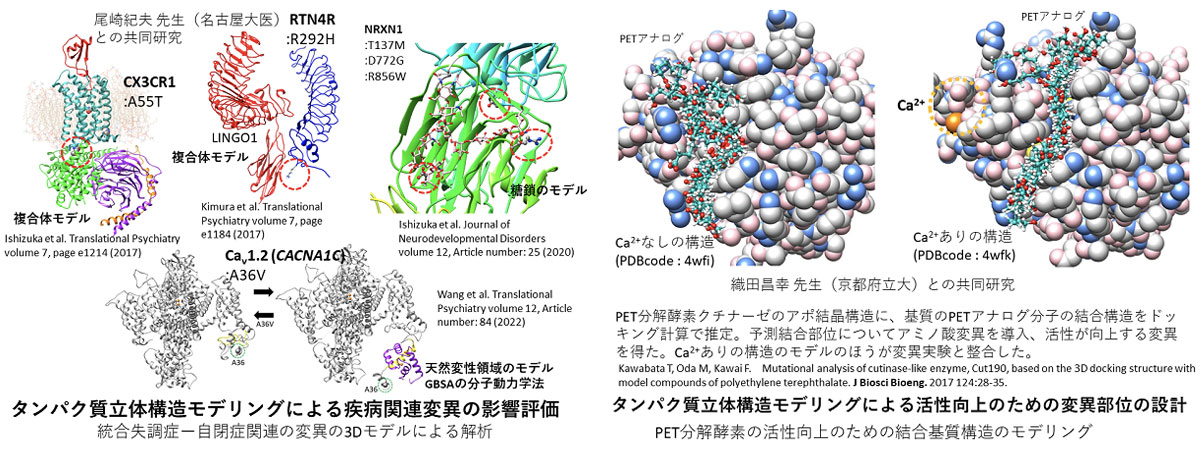
支援担当者は、これまでに、以下のような研究支援を行っている。まず、タンパク質立体構造モデリングによる疾病関連変異では、統合失調症・自閉症に関連する変異部位について、様々な立体構造モデルを作成することで、影響を受ける分子機能を予想し、実験的に確認するための仮説設計に役立てた。そのために、タンパク質複合体のモデル、糖鎖修飾されたタンパク質のモデル、天然変性領域を含むタンパク質のモデルなど様々な分子モデルを作成した。また、タンパク質立体構造モデリングによる活性向上のための変異部位の設計では、PET分解酵素について、基質と結合したタンパク質立体構造のモデリングを行い、結合部位を予測することで、変異導入実験に役立てた。
・タンパク質立体構造モデリングによる疾病関連変異の影響評価
Wang C et al. Identification of ultra-rare disruptive variants in voltage-gated calcium channel-encoding genes in Japanese samples of schizophrenia and autism spectrum disorder. Transl Psychiatry. 2022 12(1):84. doi: 10.1038/s41398-022-01851-y.
Ishizuka K et al. Functional characterization of rare NRXN1 variants identified in autism spectrum disorders and schizophrenia. J Neurodev Disord. 2020 12:25. doi: 10.1186/s11689-020-09325-2.
Kimura H et al. A novel rare variant R292H in RTN4R affects growth cone formation and possibly contributes to schizophrenia susceptibility. Transl Psychiatry. 2017 7:e1214. doi: 10.1038/tp.2017.170.
Ishizuka K et al. Rare genetic variants in CX3CR1 and their contribution to the increased risk of schizophrenia and autism spectrum disorders. Transl Psychiatry. 2017 7:e1184. doi: 10.1038/tp.2017.173.
・タンパク質立体構造モデリングによる活性向上のための変異部位の設計
Kawabata T et al. Mutational analysis of cutinase-like enzyme, Cut190, based on the 3D docking structure with model compounds of polyethylene terephthalate. J Biosci Bioeng. 2017 124:28-35. doi: 10.1016/j.jbiosc.2017.02.007.
支援担当者は、以下のタンパク質立体構造のモデリングに関連するプログラム、サーバ、データベースの開発・運営を行っている。
[複合体検索・モデリングサーバHOMCOS]
https://homcos.pdbj.org Kawabata T. HOMCOS: an updated server to search and model complex 3D structures. J Struct Funct Genomics. 2016 17:83-99. doi: 10.1007/s10969-016-9208-y.
[天然リガンド立体構造データベースNLDB]
https://nldb.sb.ecei.tohoku.ac.jp/nldb/top Murakami Y, Omori S, Kinoshita K. NLDB: a database for 3D protein-ligand interactions in enzymatic reactions. J Struct Funct Genomics. 2016 17:101-110. doi: 10.1007/s10969-016-9206-0.
[日本人マルチオミックス参照データベースjMorp]
https://jmorp.megabank.tohoku.ac.jp/202112/ Tadaka S et al. jMorp updates in 2020: large enhancement of multi-omics data resources on the general Japanese population. Nucleic Acids Res. 2021 49(D1):D536-D544. doi: 10.1093/nar/gkaa1034.
[化学構造比較プログラムKCOMBU]
https://pdbj.org/kcombu/ Kawabata T. Build-up algorithm for atomic correspondence between chemical structures. J Chem Inf Model. 2011 51:1775-87. doi: 10.1021/ci2001023.
[結合ポケット同定プログラム ghecom]
https://pdbj.org/ghecom/ Kawabata T. Detection of multiscale pockets on protein surfaces using mathematical morphology. Proteins. 2010 78:1195-1211. doi: 10.1002/prot.22639.
[電顕3D密度マップと原子モデルの重ね合わせプログラム gmfit]
https://pdbj.org/gmfit/ Kawabata T. Multiple subunit fitting into a low-resolution density map of a macromolecular complex using a gaussian mixture model. Biophys J. 2008 95:4643-58. doi: 10.1529/biophysj.108.137125.