| 所属 |
① 東京大学 定量生命科学研究所 ② 東京大学 定量生命科学研究所 |
|
|---|---|---|
| 氏名 |
① 白髭 克彦 ② 中戸 隆一郎 |
|
| AMED 事業 |
課題名 | 先端エピゲノミクス・1細胞解析支援 |
| 代表機関 | 東京大学 | |
| 代表者 | 白髭 克彦 | |
Hi-C、ChIP-seq、RNA-seq、クロマチン高次構造解析、高難度シーケンス技術支援
Hi-C及びその発展技術である Micro-C、HiChIP、MicroChIP、Pore-C等の関連技術による細胞核内のクロマチン高次構造解析支援を行う。また通常のChIP-seq、RNA-seq等のエピゲノム解析技術・シーケンス解析技術に加え、少数細胞ChIP-seq技術や、定量spike-in ChIP-seq、長鎖シーケンス技術など当研究室が保有する様々な技術を用いて、あるいは新規の手法や発展手法を用いて最適なシーケンス解析を行う。
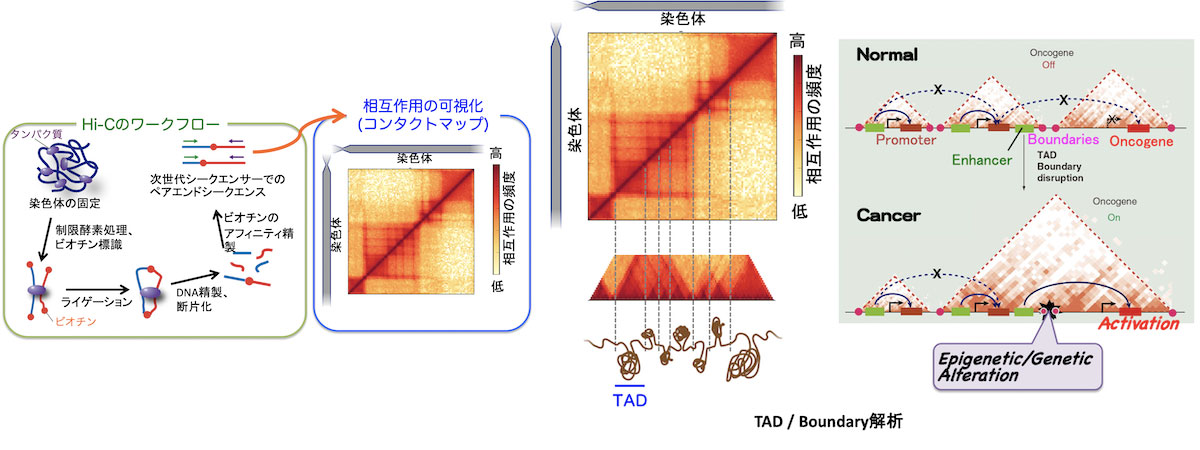
Hi-C法は、染色体上の空間的に近接して配置されている領域を同定する chromosome conformation capture (3C) の手法と次世代シークエンサー技術を組み合わせることで、染色体の高次構造を網羅的に解析する手法である。Hi-Cでは、ホルマリンで固定した細胞や組織の染色体の高次構造を、制限酵素によるDNAの断片化と、続く近位断片間でのライゲーション反応を通じて、染色体上で近接する領域を不連続な接合サイトとしてNGSで網羅的に同定し解析することができる。
Hi-Cの解像度(数十~数百 kbp)を改善した手法がMicro-Cである。ゲノムの断片化にリンカーDNAを切断する酵素MNaseを用いることで、ゲノムの断片をモノヌクレオソームにまで細分化し、検出の解像度を飛躍的に向上(~数百 bp)させた手法である。また、これらのHi-C/Micro-Cライブラリのうち、注目するタンパク質を抗体によって濃縮し、標的因子が介在する高次構造のみに限定して解析を行う手法がHiChIP/MicroChIPである。また、Hi-Cライブラリを長鎖シーケンサー(ナノポアシーケンサー)を用いて読むことで多点間での相互作用を検出する方法がPore-C法である。
本支援ではHi-C及びその発展技術であるMicro-C、HiChIP、MicroChIP、Pore-C等の関連技術による細胞核内のゲノム3次元構造(ヌクレオーム)解析支援を行う。支援は提供試料からのライブラリ調製、NGSによる配列決定、大規模解析サーバーを用いたコンタクトペアのマッピング等の1次解析から、TAD検出、コンパートメント解析、エンハンサー=プロモーター相互作用の検出といった高次データ解析までを一貫して行う。
また当研究室では通常のChIP-seq、RNA-seqなどの解析技術以外にも、支援メニューには載せきれない多くのエピゲノム解析技術を保有している。そこで支援者とのカウンセリングを通じて、我々がこれまで培ってきた少数細胞ChIP-seq技術や、spike-in ChIP-seq、small RNA-seq、長鎖シーケンス技術など様々な技術、あるいは新規の手法や発展手法を用いて、支援希望者の研究にとって最適なシーケンス技術を提案し、研究の進展を図る。やってみたいシーケンス技術がある、研究上の問題をシーケンス技術で解決したい、という方の要望に広く対応する。
・Hi-C法によるTAD構造解析、Micro-C法によるEnhancer - Promoter相互作用解析
・HiChIP、MicroChIPを用いたタンパク質に介在される高次構造の解析
・Pore-C法による多点間相互作用解析
・通常のChIP-seq、RNA-seq等に加え、少数細胞でのChIP-seqやspike-in ChIP-seq、small RNA-seq等の発展技術を利用した解析
・Nanopore sequencerを使用した長鎖シーケンス解析
・その他、さまざまなシーケンス解析技術
https://www.iqb.u-tokyo.ac.jp/chromosomeinformatics/index.html