| 所属 |
① 東京科学大学 情報理工学院 ② 東京科学大学 情報理工学院 ③ 東京科学大学 情報理工学院 |
|
|---|---|---|
| 氏名 |
① 関嶋 政和 ② 石田 貴士 ③ 大上 雅史 |
|
| AMED 事業 |
課題名 | スーパーコンピュータ資源及び大規模シミュレーションとAIに基づく創薬・生命科学の支援 |
| 代表機関 | 東京科学大学 | |
| 代表者 | 関嶋 政和 | |
スーパーコンピュータ、機械学習、バーチャルスクリーニング、分子動力学シミュレーション
東京工業大学のスーパーコンピュータTSUBAME上での計算資源の提供及び大規模シミュレーションと機械学習に基づく創薬・生命科学の支援を行うことを目的とする。具体的には、(A)共用ファシリティとしての東工大TSUBAMEスパコンの創薬支援分野における利活用支援、(B)インシリコスクリーニングおよび標的タンパク質と低分子化合物などとの相互作用様式決定支援、(C)中分子モダリティに関する支援、(D)立体構造予測に基づく天然変性領域予測と複合体モデリングに関する支援、(E)実験研究者のデータから新しい知見を見いだす機械学習・データサイエンス支援、を行う。 (A) TSUBAME3は導入時世界13位の計算性能、世界4位の大規模データ処理性能と高い性能を示し、令和5年導入予定のTSUBAME4に更新予定である。 (B)ドッキングと機械学習を組合せた相互作用様式の推定技術(Yasuo et al. JCIM, 2019(表紙採用); 特願2019-015086)に強みがあるが、様々なプロジェクトにおいてヒット化合物を発見した実績を有している。(C)深層学習によるペプチド血漿タンパク質結合率推定(Li et al. Bioinformatics, 2021; 特願2021-35648)は現状の最高精度を示しており、大規模MD計算によるペプチド膜透過性予測(Sugita et al. JCIM, 2021(表紙採用); 特願2021-23750)は100ペプチド以上の膜透過性予測を世界で初めて実現している。(D)立体構造予測CASPコンテストにて世界最高精度を達成した天然変性領域予測PrDOS (Ishida et al. NAR, 2007)や、網羅的タンパク質間相互作用(PPI)予測MEGADOCK (Ohue, et al. Bioinformatics 2014)など、それぞれが世界最高水準の精度でかつ高い実績を有している。MEGADOCKによる新規PPI発見の例も複数存在する (Kami, et al. PLoS ONE 2018等)。(E)様々な製薬企業との共同研究経験に基づく、実験データに対するオンデマンド解析に特色がある

スーパーコンピュータTSUBAME3を用いた分子動力学シミュレーションにより、新型コロナウイルス(SARS-Cov-2)感染症(COVID-19)の治療薬探索に重要となる、ウイルス複製に必要な酵素の一つであるMain Protease(Mpro、メインプロテアーゼ)もしくは3C like Protease (3CL Protease)の複製機能阻害をする薬候補化合物が満たすべきファーマコフォアのモデリングに成功した。本研究によりスーパーコンピュータTSUBAME3を用いることで1マイクロ秒の分子動力学(MD)シミュレーションを実施し、SARS-CoV-2 Mproと3つの薬剤候補との重要な相互作用を明らかにし、ファーマコフォアのモデリングに成功した。本研究では、SARS-CoV-2 Mproの41番目のHis(ヒスチジン)、143番目のGly(グリシン)、166番目のGlu(グルタミン酸)は、すべてのMDシミュレーションにおいて共通する官能基と相互作用を形成していた。これらの相互作用は、SARS-CoV-2 Mproに対する薬剤の重要なターゲットであることを示唆している。
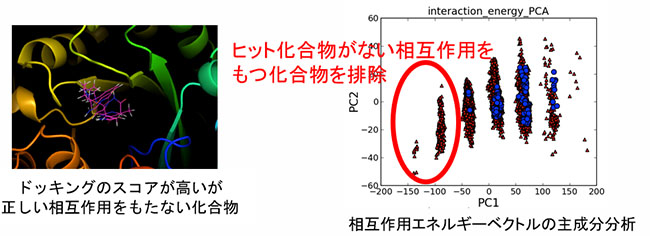
また、Main Proteaseの阻害化合物を新規に6個発見した。ペプチド様化合物とは異なる化合物空間からヒット化合物を取得するために、まず化合物ライブラリからペプチド結合を有する2級アミド化合物を取り除いてから、ドッキングシミュレーションによるSBVS(立体構造に基づくバーチャルスクリーニング)を実施した。SBVSによって抽出した180化合物を対象に酵素阻害アッセイ実験を実施したところ、6化合物でIC50の測定に成功した。
関嶋政和:http://www.cbi.c.titech.ac.jp/
石田貴士:http://www.cb.cs.titech.ac.jp/
大上雅史:https://www.li.c.titech.ac.jp/