| 所属 |
① 東京大学 大学院農学生命科学研究科 ② 東京科学大学 難治疾患研究所 |
|
|---|---|---|
| 氏名 |
① 寺田 透 ② 森脇 由隆 |
|
| AMED 事業 |
課題名 | 分子シミュレーションによる生体高分子の機能の予測と解析 |
| 代表機関 | 東京大学 | |
| 代表者 | 寺田 透 | |
ドッキングシミュレーション、複合体モデリング、分子動力学(MD)シミュレーション、自由エネルギー計算
タンパク質-タンパク質、タンパク質-核酸、タンパク質-低分子化合物の複合体構造に対して、分子動力学(MD)シミュレーションを行い、分子間相互作用を解析する。また、必要に応じて、結合自由エネルギーの計算や、変異による複合体構造や、結合自由エネルギーの変化の解析を行う。
タンパク質-タンパク質、タンパク質-核酸、タンパク質-低分子化合物の複合体構造予測を行う。タンパク質-タンパク質の複合体構造予測では、まず、AlphaFoldを用いた予測を行う。十分な予測信頼度が得られない場合は、ドッキングシミュレーションを行う。タンパク質-核酸、タンパク質-低分子化合物の場合は、ドッキングシミュレーションにより、複合体構造予測を行う。ドッキングシミュレーションにより適切な複合体モデルが得られない場合は、マニュアルでモデリングを行う。得られた複合体モデルに対して、MDシミュレーションを行い、モデルの精密化と安定性の評価を行う。安定性が十分でない場合は、モデルを修正し、再びMDシミュレーションを行う。これを安定なモデルが得られるまで繰り返す。また、必要に応じて結合自由エネルギーの計算を行う。
タンパク質SRKとタンパク質SP11の複合体構造予測と、結合自由エネルギーの計算を行った。アブラナ科植物では、雌ずい側因子の受容体キナーゼSRKと花粉側因子のSP11が、同じハプロタイプ由来のとき、特異的に結合してSRKが活性化し、自家不和合性反応が起こると考えられている。本研究ではまず、ハプロタイプ8と9のSRK/SP11複合体の結晶構造をもとに、立体構造未知の22種類のSRK、9種類のSP11の立体構造予測を行った。SP11の立体構造予測では、後述の高度化手法を用いた。結晶構造が得られている2種類を加えた、24種類のSRK、11種類のSP11を、配列類似性に基づいて2つのクラス(クラスI、II)に分け、それぞれのクラスで、総当たり(クラスⅠ:SRK 20種類×SP11 7種類、クラスII:SRK 4種類×SP11 4種類)で複合体モデルを作成した。ただし、結晶構造が得られているペアについて結晶構造を複合体モデルとして利用した。続いて、それぞれの複合体モデルについて、MM-GBSA法を用いて結合自由エネルギーΔGを計算した。この結果、同じハプロタイプが複合体を形成しているとき、ΔGは特に大きな負の値を示すことが明らかとなり、提唱されている自家不和合性反応のメカニズムを支持する結果が得られた [Murase et al., Nat. Commun. 11, 4916 (2020)]。
植物ホルモン・ブラシノステロイドの情報伝達系で機能する転写因子BIL1と、これが認識するパリンドローム配列CACGTGの相互作用を解析した。同じDNA配列を認識する別の転写因子MYC2は両端のCAおよびTGを置換すると親和性が大きく低下するのに対して、BIL1では低下の程度が小さいことが知られている。本研究ではまず、BIL1・DNA複合体、MYC2・DNA複合体に対して 1 μsのMDシミュレーションを行った。続いて、両端の2塩基CAを、TAまたはCGに1塩基ずつ置換する自由エネルギー摂動計算を行った。DNA単体についても同様の計算を行い、塩基置換に伴う結合自由エネルギーの変化を計算した。この結果、実験から示唆されていた通り、両端の2塩基を置換するとMYC2に対する親和性が大きく低下するのに対し、BIL1に対しては低下の程度が小さいことが確かめられた [Nosaki et al., Sci. Rep. 11, 3879 (2021)]。
tRNAを標的とするリボヌクレアーゼとtRNAの複合体構造予測を行った。Colicin Dは抗菌活性を持つバクテリオシンの1つであり、特定のtRNAを切断して翻訳を阻害することで、殺菌作用を発揮する。Colicin Dの結晶構造と、変異体実験などにより特定された、活性に重要な残基の情報に基づき、colicin DとtRNAの複合体の立体構造の予測を行った。ここでは、ソフトウェアFTDockを用いた、形状の相補性や静電相互作用に基づくドッキングに加えて、マニュアルでのドッキングを行った。FTDockでは多数の候補構造から、tRNA切断反応のメカニズムから想定される、colicin Dの活性中心の原子と、これと相互作用するtRNAの原子が近くなるものを複数選択した。マニュアルでのドッキングでは、この2つの原子が近くなるようtRNAをcolicin Dに対して配置した後、tRNAと衝突するcolicin Dのアミノ酸の側鎖のコンフォメーションを変更した。それぞれのモデルについてMDシミュレーションを実施し、複合体構造の安定性を評価した。この結果、マニュアルドッキングで作成したモデルの安定性が最も高く、反応に必要な原子配置も維持されることが明らかとなった。さらに、MDによって精密化されたモデル構造を用いて、実験で活性に重要とされた残基の役割を議論した [Ogawa et al., RNA Biol. 18, 1193 (2021)]。
リプログラミング因子Klf4の、DNAとの親和性を高めるL507A変異による分子間相互作用の変化を解析した。Klf4とDNAの複合体の結晶構造を用いて、Klf4変異体とDNAの複合体のモデルを作成した。野生型、変異体に対してそれぞれ2 μsのMDシミュレーションを、初速を変えて3回ずつ行った。立体構造分布を解析したところ、変異体には野生型に見られない構造が現れることが示された。この構造では、Klf4のDNAとの間に新たな塩橋や水素結合が形成されており、これらによってDNAとの親和性が高まっていることが示唆された [Borisova et al., iScience, 25, 103525 (2022)]。
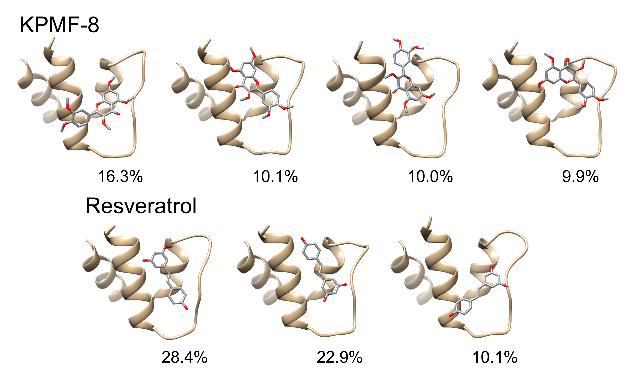
Sirtuin 1(SIRT1)とこれを活性化する低分子化合物との相互作用を解析した。SIRT1は核内に局在し、NAD+を利用してアセチル化されたリシン残基からアセチル基を除去する反応を触媒する。SIRT1は加齢による病気(2型糖尿病、神経変性、ガン、アルツハイマー病など)を防ぐ働きがあることが知られている。これまで、resveratrolがSIRT1を活性化することが知られていたが、2014年に黒ウコンから同定されたKPMF-8が同様の活性を持つことが示された。等温滴定カロリメトリー(ITC)とNMRにより、resveratrolとKPMF-8は、いずれもSIRT1のN端ドメイン(NTD)に結合することが示されたため、ドッキングシミュレーションによりNTDとKPMF-8の複合体構造を予測した。MDシミュレーションを用いて、このモデルの安定性の評価を行ったところ、いずれの化合物もNTDへの結合構造には複数の安定構造があり、その間を速いタイムスケール( 1 μs)で交換していることが明らかとなった。これは、NMRの化学シフト変化の実験結果と矛盾しない結果である [Zhang et al., Commun. Biol. 4, 209 (2021)]。
バイオインフォマティクス、立体構造予測、分子動力学シミュレーション、量子化学計算などを駆使して、タンパク質の機能発現のメカニズムを明らかにするための研究を行っている。
研究室ホームページ:http://www.bi.a.u-tokyo.ac.jp/