| 所属 |
① 東京科学大学 難治疾患研究所 ② 東京大学 大学院農学生命科学研究科 |
|
|---|---|---|
| 氏名 |
① 森脇 由隆 ② 寺田 透 |
|
| AMED 事業 |
課題名 | 分子シミュレーションによる生体高分子の機能の予測と解析 |
| 代表機関 | 東京大学 | |
| 代表者 | 寺田 透 | |
量子化学計算、QM/MM法、遷移状態、反応経路、分子動力学(MD)シミュレーション
酵素と基質の複合体構造をもとに、量子化学計算により、酵素の触媒機構を解析する。ここでは、計算コストを削減するために、酵素の活性中心を構成するアミノ酸残基と基質の原子を量子力学(quantum mechanics; QM)で、それ以外の原子を分子力学(molecular mechanics; MM)で扱う、QM/MM法を用いる。酵素・基質複合体に対して分子動力学(MD)シミュレーションを行い、複合体構造の安定性を確認した後、QM/MM法を用いてエネルギー最小化を行う。続いて想定される反応経路に沿って原子を動かし、ポテンシャルエネルギー曲面を求める。ポテンシャルエネルギーが極大となる構造に対して、遷移状態の最適化を行う。活性化エネルギーの妥当性を検討し、問題がある場合は、複合体構造および反応経路の再検討を行う。また、量子化学計算を用いて、鉄など金属イオンの配位構造の解析を行う。
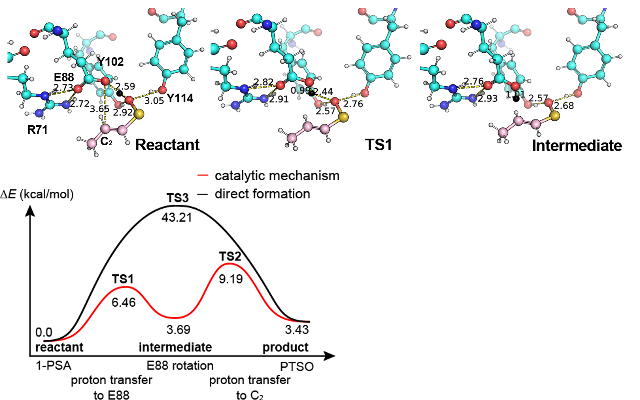
タマネギに存在する催涙因子合成酵素(lachrymatory factor synthase; LFS)の触媒機構の解析を行った。LFSは、前駆体(E)-1-propensulfenic acid(1-PSA)を催涙因子 (Z)-propanthial S-oxide(PTSO)に転化する酵素である。しかし、1-PSAが不安定なために、長らくLFSの触媒機構は不明であった。本研究では、LFSの結晶構造に基質をドッキングし、MDシミュレーションを用いて安定な複合体構造を探索した。ここから、反応に有利な構造を取り出し、QM/MM法を用いて反応のエネルギー地形を計算し、遷移状態と反応経路を決定した [Arakawa et al., ACS Catal. 10, 9-19 (2020)]。
グラム陰性細菌Thermus thermophilus由来のタンパク質TtFbpAが結合する鉄イオンの配位構造を解析した。TtFbpAは鉄取込みに関わる鉄結合タンパク質で、pH 7.5では5配位、pH 5.0の6配位の構造をとる。我々は、2つの構造について量子化学計算を行い、リガンドの分子種とプロトン化状態を決定した。また、pH 7.5の5配位の構造より、pH 5.0の6配位の構造の方が、鉄結合強度が弱いことが知られているが、我々は、鉄結合部位周辺のタンパク質構造に違いがあることを見出し、配位形式の違いに加えてタンパク質構造の違いが鉄結合強度の違いをもたらしていることを明らかにした [Lu et al., Metallomics, 11, 2078-2088 (2019)]。
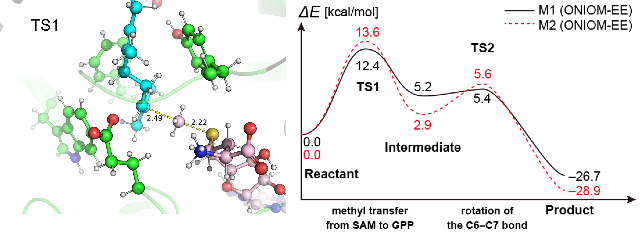
放線菌のbenzastatinの生合成にかかわる酵素BezAの触媒機構の解析を行った。この酵素は、S-adenosylmethionine(SAM)のメチル基をgeranyl diphosphate(GPP)のC-6位に転移する反応を触媒する。ここではまず、BezAと、SAMがメチル基を失った生成物であるS-adenosyl-l-homocysteine(SAH)との複合体の結晶構造をもとに、BezA・GPP・SAMの3者複合体のモデルを構築した。MDシミュレーションを用いてモデルを精密化した後、QM/MM計算を行い、反応経路と遷移状態を決定した [Tsutsumi et al., Angew. Chem. Int. 61, e202111217 (2022)]。
バイオインフォマティクス、立体構造予測、分子動力学シミュレーション、量子化学計算などを駆使して、タンパク質の機能発現のメカニズムを明らかにするための研究を行っている。
研究室ホームページ:http://www.bi.a.u-tokyo.ac.jp/