| 所属 | ① 早稲田大学 理工学術院 | |
|---|---|---|
| 氏名 | ① 由良 敬 | |
| AMED 事業 |
課題名 | 1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援 |
| 代表機関 | 早稲田大学 | |
| 代表者 | 由良 敬 | |
データベース、ホモロジーモデリング、タンパク質機能予測、データ発信用ウェブ開発
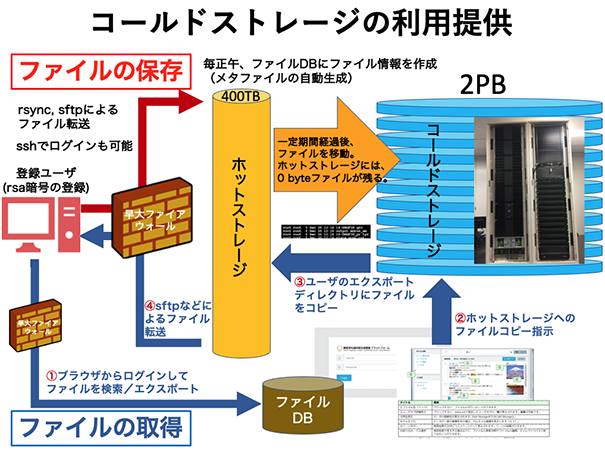
・多くの数値/文字データのコールドストレージにおける長期保存
近年の生命科学研究では、大量の数値データと文字データをあつかうようになった。これらのデータを用いて結果を得た後も、そのデータの保存が求められるようになってきた。ところが手元に大きなストレージがない場合には、データの保存が非常に難しいのが実態である。そこで本支援では、データ解析のみならず、そのデータの長期保存を実施する。早大に設置したコールドストレージは、データをBluRayに書き込むため、初期データからの改変なく長期保存ができると共に、必要な際にはネットワークを介して読み出すことも可能である。
オミックスデータが得られた際に、それらのデータをデータベース化することと、タンパク質の配列/構造/機能の予測を支援する。
・シロイヌナズナのSAP130とCSN1の相互作用構造を推定した(Shiori S. Aki, Kei Yura, Takashi Aoyama, Tomohiko Tsuge (2021) Journal of Plant Research, 134, 279-289.)。
・ABCトランスポートの多数の立体構造情報と遺伝子のミスセンス情報を組み合わせることで、ミスセンス変異がタンパク質の動的構造の要部分に存在することを見いだした(Sakamoto M., Suzuki H., Yura, K. (2019) Biophysics and Physicobiology, 16, 68-79.)。
・神経細胞で発現する遺伝子情報を解析し、細胞の形態変化に関与する遺伝子候補を抽出した。その結果に基づく実験により、ほ乳類脳の層構造形成に関与する遺伝子群を特定した(Ohtaka-Maruyama C., ... Yura, K., ... Maeda, N. (2018) Science, 360, 313-317.)。
・ヒトデのアポトーシス関連タンパク質の立体構造をモデリングし、ほ乳類のアポトーシス機構との構造的および機能的差異を推定した(Tamura, R., ... Yura, K., Chiba, K. (2018) Scientific Reports, 1611.)。
・Autophagy関連タンパク質を収集したデータベースを公開開始した(Mizutani, H., Sugawara, H., ... Yura, K. (2017) BMC Structural Biology, 17, 4.)。