| 所属 |
① 早稲田大学 理工学術院 ② 早稲田大学 理工学術院 |
|
|---|---|---|
| 氏名 |
① 大森 聡 ② 由良 敬 |
|
| AMED 事業 |
課題名 | 1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援 |
| 代表機関 | 早稲田大学 | |
| 代表者 | 由良 敬 | |
分子動力学シミュレーション、モデリング、ドッキング、粗視化モデル、線形応答理論
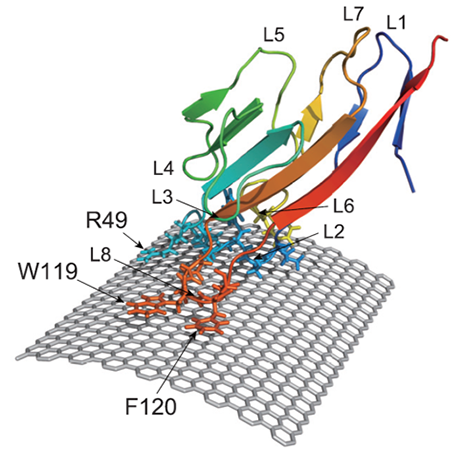
分子動力学シミュレーションによるタンパク質-カーボンナノチューブ複合体の構造予測。
結合活性に影響を与える新規の変異を支援者に提案。
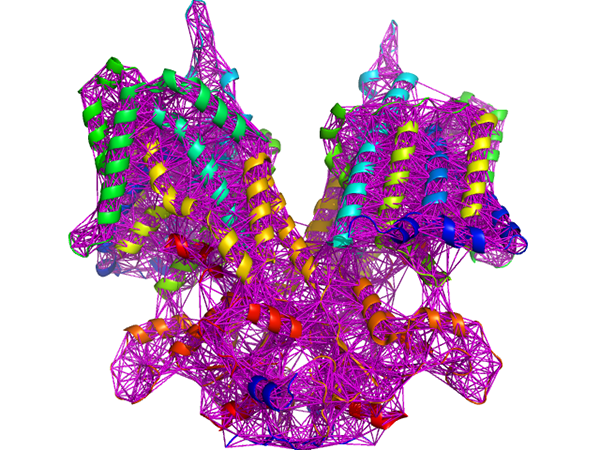
弾性ネットワークモデルによる簡易的なタンパク質の構造変化解析
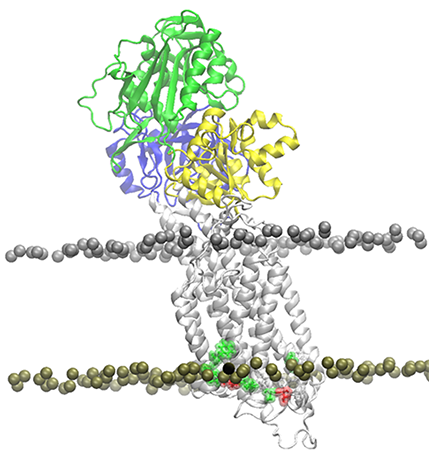
野生型と変異型のシミュレーションを比較することでタンパク質の機能発現機構を推定
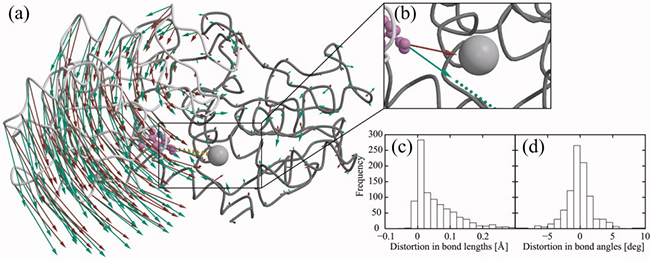
線形応答理論(LRT)によるリガンド結合に伴う構造変化の予測。
(a)予測構造変化(青色矢印)は実験で解かれた構造変化(茶色矢印)とよく合致。
(b)リガンド結合は活性部位にはたらく引力として簡易化。
タンパク質モデリング、タンパク質-タンパク質およびタンパク質-リガンドドッキング、分子動力学シミュレーションを組み合わせ、タンパク質の動的な機能発現機構の解析を行う。特にタンパク質分子動力学シミュレーションによるタンパク質運動および機能発現機構の探索については詳細な解析が可能。また、分子動力学シミュレーションで追従できるタンパク質の運動は数μ秒程度が限界だが、線形応答理論、粗視化モデルなどを応用することにより、より長いタイムスケールのタンパク質構造変化を予測することも可能。支援申請者の要望に応じてこれらの技術や理論を柔軟に組み合わせて、タンパク質の機能推定、ウェット実験への提案などを行う。